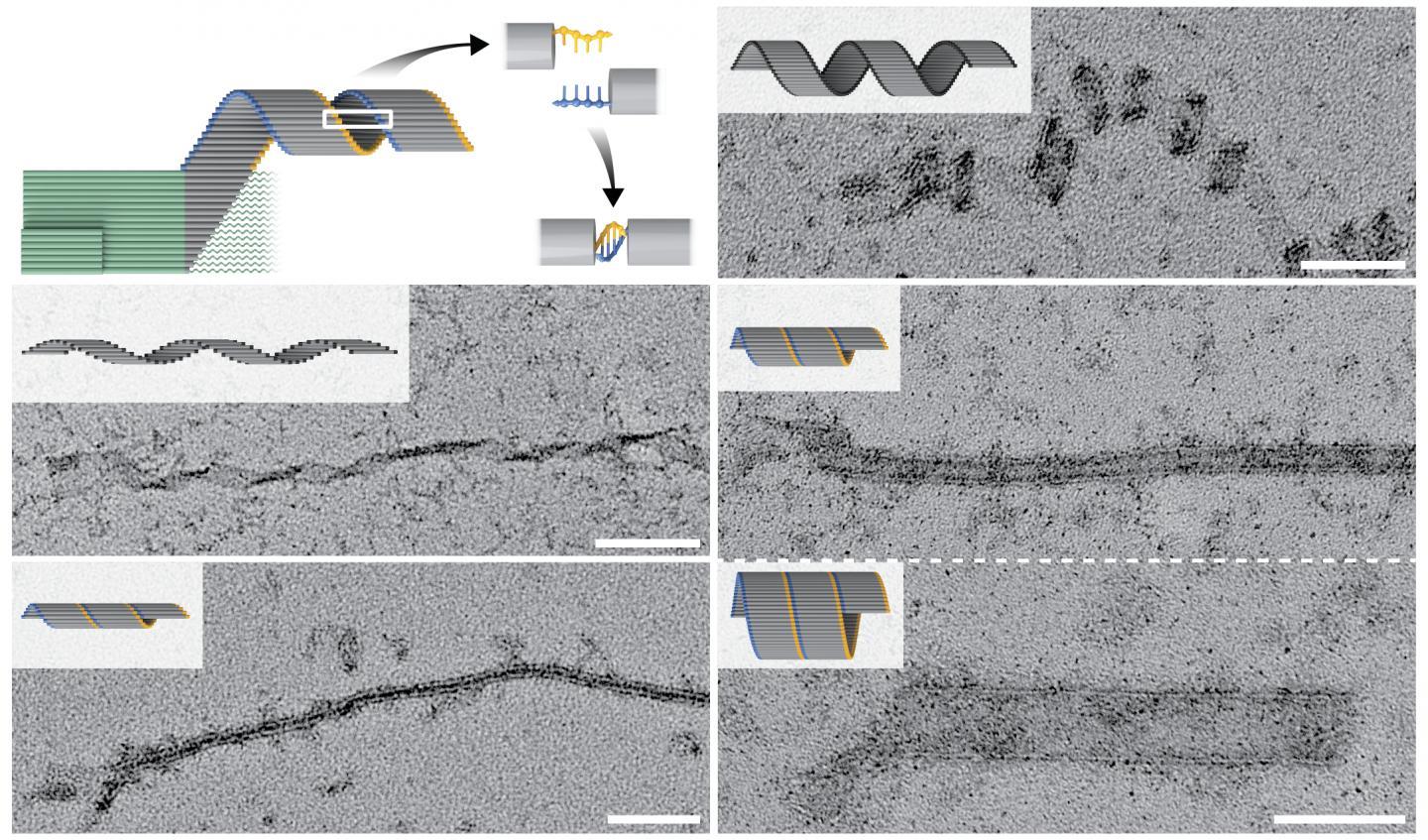
(波士顿)-由Wyss创办核心学院成员William Shih博士领导的哈佛大学Wyss生物启发工程研究所和达纳-法伯癌症研究所(DFCI)的纳米生物技术专家团队设计了一种可编程的DNA自组装技术。该策略解决了强大的成核控制的关键挑战,并为超灵敏诊断生物标志物检测和具有纳米尺寸特征的微米尺寸结构的可扩展制造等应用铺平了道路。使用这种称为“交叉聚合”的方法,研究人员可以通过严格依赖种子的成核作用,从细长的DNA单链(称为“板条”)开始编织纳米带。该研究发表在《自然通讯》上。
DNA纳米结构具有高度的生物相容性和可编程性,因此具有解决各种诊断,治疗和制造难题的巨大潜力。为了发挥有效的诊断设备的作用,例如,DNA纳米结构可能需要通过触发与在医疗点或临床实验室环境中可使用的低成本仪器兼容的扩增读数,来对目标分子的存在做出特异性反应。
大多数DNA纳米结构是使用两种主要策略之一组装的,每种策略都有其优势和局限性。“ DNA折纸”由长的单链支架链形成,该支架链由许多较短的短链稳定在二维或三维构型中。它们的组装严格取决于脚手架链,导致坚固的全有或全无折叠。尽管它们可以在宽范围的条件下以高纯度形成,但是它们的最大尺寸受到限制。另一方面,“ DNA积木”可以通过许多短的模块化链组装更大的结构。但是,它们的组装需要严格控制的环境条件,可以在没有种子的情况下以假方式启动,并且会产生很大一部分不完整的结构,需要将其清除掉。
“在过去的二十年中,DNA折纸的引入一直是DNA纳米技术领域最具影响力的进步。我们在这项研究中开发的纵横交联方法建立了将可控制的DNA自组装扩展到许多其他基础之上Wyss的分子机器人计划的共同负责人,同时也是哈佛医学院和DFCI教授的Shih说道。“我们预想,在需要极高灵敏度的诊断应用中,纵横交联聚合将广泛地实现具有可寻址纳米级特征,算法自组装和零背景信号放大的二维或三维微观结构的全有或全无的形成。”
 生物帮
生物帮